Étudier la dynamique d’expression du protéome pour guider les expériences en biologie cellulaire
Dans le cadre du projet ANR Dynotep, les équipes du CEA, notamment les laboratoires EDyP et Odycell, ont mené une étude visant à mieux comprendre les dynamiques d’expression protéique lors du processus d’endocytose chez Dictyostelium discoideum, un organisme modèle couramment utilisé en biologie cellulaire. Ce processus, analogue à une digestion intracellulaire, est déclenché lorsque l’amibe prélève des éléments nutritifs dans son environnement. L’objectif scientifique était de caractériser les profils d’expression des protéines impliquées dans ce mécanisme, à différentes étapes temporelles, afin d’identifier des trajectoires caractéristiques.
Au-delà de la connaissance biologique, l’un des enjeux majeurs de ce travail était d’ordre méthodologique : comment réduire le nombre d’expériences nécessaires en laboratoire sans perdre en précision ? L’idée était d’utiliser des techniques d’analyse exploratoire (exemple: le calcul du z score) pour repérer des groupes de protéines aux dynamiques similaires, et ainsi focaliser les efforts expérimentaux sur un sous-ensemble représentatif. Ce type d’approche, s’il est fiable et reproductible, permet de limiter les coûts, d’accélérer les protocoles et de concentrer les validations sur les cibles les plus prometteuses.
Des données protéomiques complexes à structurer
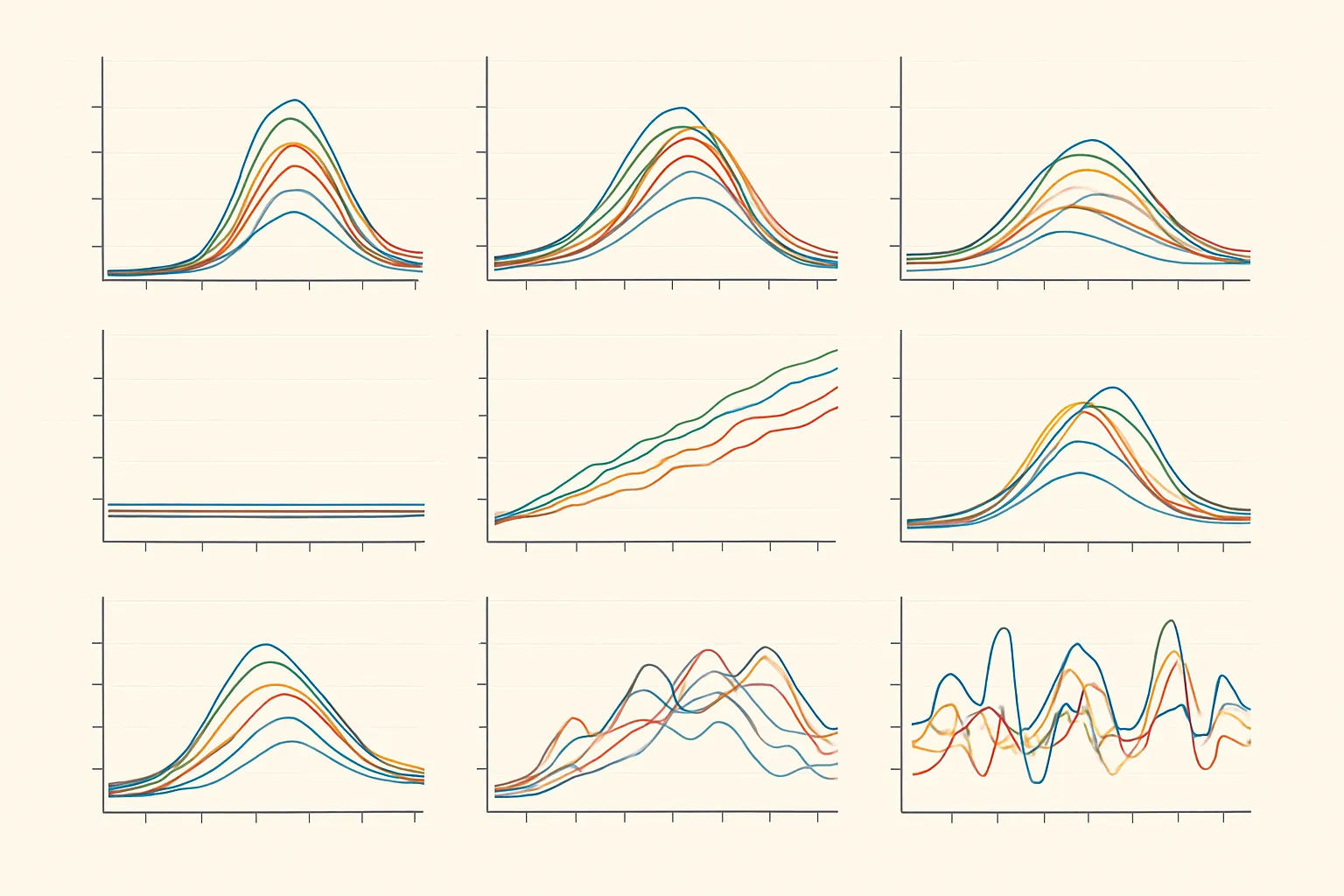
Le jeu de données analysé provient de mesures de spectrométrie de masse, produites à différentes étapes d’un protocole expérimental standardisé. Chaque protéine est représentée par une série temporelle de valeurs quantitatives, correspondant à son abondance au fil du temps. Ces données, riches et complexes, comportent plusieurs centaines de profils distincts, dont les dynamiques ne sont pas facilement comparables sans un outillage statistique et visuel adapté.
Chaque protéine est associée à des variables complémentaires : des propriétés biochimiques, des mesures de variation temporelle, ou encore des valeurs normalisées (calcul du z score) permettant une comparaison indépendante des niveaux d’expression absolus. Le défi consistait à structurer ces informations de manière à identifier des régularités dans les trajectoires, en détectant des profils redondants ou, au contraire, singuliers.
Résultats obtenus : des regroupements éclairants
L’analyse, incluant le calcul du z score, a permis de regrouper les protéines selon des comportements temporels similaires, révélant ainsi l’existence de trajectoires d’expression typiques. Ces regroupements ont été visualisés sous forme d’arbres hiérarchiques empilés (Stacked Trees), facilitant la navigation entre les groupes, l’examen des profils internes et la comparaison des dynamiques. Les biologistes ont pu annoter certains de ces groupes, en sélectionner d’autres comme représentatifs, et identifier des « signatures » pouvant guider les expériences de validation.
Cette segmentation raisonnée a permis de réduire significativement le nombre de protéines à suivre expérimentalement. Le protocole a ainsi gagné en efficacité, tout en conservant une couverture représentative des dynamiques observées. En plus du gain de temps, cette approche a permis d’engager une discussion plus fine entre statisticiens, bioinformaticiens et biologistes sur la pertinence des profils repérés.
Apport de Treensight : unifier l’analyse
Treensight Explorer a offert un environnement unifié pour l’analyse des données temporelles complexes, sans rupture entre la phase de préparation, d’analyse, de visualisation et d’interprétation. L’intégration fluide des variables biochimiques, temporelles et quantitatives a permis d’explorer les profils protéiques de manière intuitive, tout en maintenant une rigueur méthodologique complète.
Les visualisations hiérarchiques, combinées aux statistiques dont le calcul du z score, ont permis de passer d’un grand nombre de séries individuelles à des représentations structurées et synthétiques. La capacité à documenter chaque étape d’analyse, à annoter les groupes détectés et à exporter des rapports lisibles a grandement facilité le dialogue entre les disciplines. Cette approche a convaincu les chercheurs qu’un outil bien pensé pouvait faire le lien entre complexité biologique et exploration méthodique, sans exiger de manipulations techniques complexes.
Ce use case reste un exemple emblématique de la manière dont Treensight peut contribuer à des projets de recherche fondamentale, en s’adaptant à la fois aux contraintes expérimentales et aux exigences scientifiques.